收集一堆生物分子三维结构浏览软件
Jun 03, 2008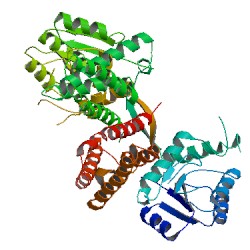
上图是某海洋细菌Synechococcus elongatus的生物钟蛋白质KaiA的分子结构。 可惜这幅图是二维的,如果想从其他角度看这个蛋白质分子,就需要三维结构浏览器啦。
首先有一堆网页版的浏览器,比如KiNG, JMol, 这些软件基于web浏览器,不用本地安装,但是都需要Java支持。在http://www.rcsb.org/pdb/explore.do?structureId=1r8j的右侧图片下面有”Display Options“,点击下面的各个brower就可以使用了。
以下介绍本地版的分子三维结构浏览器。
RasMol
RasMol是最著名的分子三维结构浏览器,由Roger Sayle在1992年开发,之后易手数人进行维护更新。免费开源,可以用鼠标旋转观看,可以以多种模式显示(如球棍模式,空间填充模式,卡通模式等),并可以导出为平面图形文件。
下载在这里,有Windows版本和源代码。Ubuntu可以直接 sudo apt-get install rasmol
下面就用上述蛋白质演示一下。
首先在这个页面点下载图标,下载该蛋白质的pdb文件。然后打开RasMol,Ubuntu下自动安装在Applications->Education目录下。或者直接在终端输入rasmol运行。然后打开下载的1R8J.pdb文件,开始胡搞。Display菜单可以选择显示模式,Colours菜单可以选择着色方式,Options菜单可以选择是否显示氢原子、是否显示镜像等。然后Export菜单允许用户把图像导出为平面图形文件。
比如下图是根据温度着色的KaiA分子结构的卡通显示模式。

此图和本文最前面的图显示的是同一个分子的结构,但因为把分子旋转了一下,所以视角不同了。
ICM Browser
http://www.molsoft.com/icm_browser.html
是Molsoft公司开发的免费三维结构浏览软件,可以直接从网上获取分子结构数据,不需要自行下载数据。也提供一些显示选项,但不如RasMol丰富。此外有自动旋转和摇摆显示功能。wikipedia上的介绍比较详细。 下载在这里,如果是Linux下运行,需要在终端输入:安装目录/icmbrowserpro -g,比如我的Ubuntu上是/home/azalea/icm-browser-pro-3.5-1n/icmbrowserpro -g
Cn3D
http://www.ncbi.nlm.nih.gov/Structure/CN3D/cn3d.shtml
没用过,据说名字的意思是See in 3D。以下完全抄自http://bio.med.stu.edu.cn/download/soft.htm
“观看蛋白质三维结构的软件,是由NCBI开发的。其设计的主要目的是为NCBI在其站点中的蛋白质结构数据库MMDB提供专业的结构观察软件。与其他的类 似的软件,如Rasmol,Weblabview等相比,其在结构观察方面主要功能上基本相似,但是图形格式上比Rasmol和Weblabview要差 一些。而在与网络连接上,该软件能依托NCBI所建立的所建立的MMDB结构数据库,能直接根据输入的序列号从数据库中利用其内嵌的Entrez搜索引擎 调出蛋白结构来进行观察,比其他软件要简便。而Cn3D主要的特点是能够将两个蛋白放在一起直观地进行三维结构上的比较,如下图中是将两种核酸外切酶的三 维结构通过VAST对准得到的结构比较图:同样,Cn3D在结构比较方面也能利用其内嵌的Blast搜索引擎直接访问Genbank数据库找到具有局部相 似性的结构数据,并在三维结构图中显示出二者具有相似性的结构区域。”
其他一些流行的分子三维结构浏览软件:
PyMol
貌似可以作为python一个模块,通过python操作。可以参考这里。
VMD
http://www.ks.uiuc.edu/Research/vmd
Protein Explorer
http://www.umass.edu/microbio/rasmol/index.html
都不了解,就不多说了,希望用过的人评价一下。